1. 前期准备
知识图谱一般用到的数据库是neo4j
neo4j可以从官网中下载,但官网链接下载需要注册账号,嫌麻烦的也可以从这里下载
neo4j下载后解压即可使用,网上对于java环境的安装和使用有很多教程,这里不多加阐述。
neo4j还需要依赖java环境,网上对于java环境的安装也有很多教程,这里也不多加阐述,这里需要注意neo4j的版本和jdk版本的兼容关系。
下面使用py2neo这个python包对neo4j数据库进行操作,这里先导入要用到的package
import os
import json
from py2neo import Node, Graph
from tqdm import tqdm这里也需要注意py2neo的版本与neo4j的版本的兼容关系
2. 数据处理
现有一份医疗相关的数据集,该数据集由若干个json组成,其中一个数据样例如下:
{
"_id": {
"$oid": "5bb578b6831b973a137e3ee6"
},
"name": "肺泡蛋白质沉积症",
"desc": "肺泡蛋白质沉积症(简称PAP),又称Rosen-Castle-man-Liebow综合征,是一种罕见疾病。该病以肺泡和细支气管腔内充满PAS染色阳性,来自肺的富磷脂蛋白质物质为其特征,好发于青中年,男性发病约3倍于女性。",
"category": [
"疾病百科",
"内科",
"呼吸内科"
],
"prevent": "1、避免感染分支杆菌病,卡氏肺囊肿肺炎,巨细胞病毒等。\n2、注意锻炼身体,提高免疫力。",
"cause": "病因未明,推测与几方面因素有关:如大量粉尘吸入(铝,二氧化硅等),机体免疫功能下降(尤其婴幼儿),遗传因素,酗酒,微生物感染等,而对于感染,有时很难确认是原发致病因素还是继发于肺泡蛋白沉着症,例如巨细胞病毒,卡氏肺孢子虫,组织胞浆菌感染等均发现有肺泡内高蛋白沉着。\n虽然启动因素尚不明确,但基本上同意发病过程为脂质代谢障碍所致,即由于机体内,外因素作用引起肺泡表面活性物质的代谢异常,到目前为止,研究较多的有肺泡巨噬细胞活力,动物实验证明巨噬细胞吞噬粉尘后其活力明显下降,而病员灌洗液中的巨噬细胞内颗粒可使正常细胞活力下降,经支气管肺泡灌洗治疗后,其肺泡巨噬细胞活力可上升,而研究未发现Ⅱ型细胞生成蛋白增加,全身脂代谢也无异常,因此目前一般认为本病与清除能力下降有关。",
"symptom": [
"紫绀",
"胸痛",
"呼吸困难",
"乏力",
"毓卓"
],
"yibao_status": "否",
"get_prob": "0.00002%",
"get_way": "无传染性",
"acompany": [
"多重肺部感染"
],
"cure_department": [
"内科",
"呼吸内科"
],
"cure_way": [
"支气管肺泡灌洗"
],
"cure_lasttime": "约3个月",
"cured_prob": "约40%",
"cost_money": "根据不同医院,收费标准不一致,省市三甲医院约( 8000——15000 元)",
"check": [
"胸部CT检查",
"肺活检",
"支气管镜检查"
],
"recommand_drug": [],
"drug_detail": []
}2.1. 设计scheme
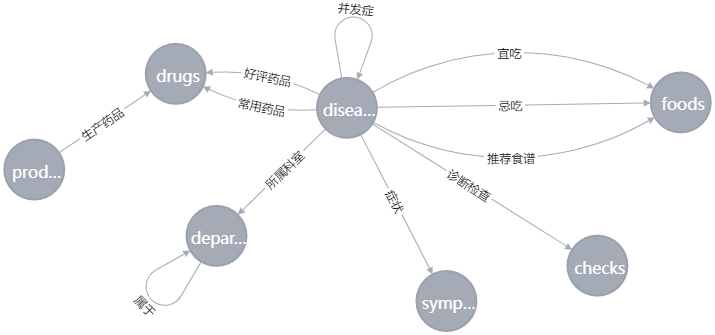
根据数据集,可以设计出如下的scheme:
2.2. 定义实体
首先定义中心实体集 diseases
# diseases节点属性
diseases_nodes_attr = ['name', 'desc', 'prevent', 'cause', 'get_prob', 'easy_get',
'cure_department', 'cure_way', 'cure_lasttime', 'symptom', 'cured_prob']
# diseases节点集合
diseases_nodes = []定义其他实体集
nodes_set_dict = {
'drugs' : set(), # 药品
'producers' : set(), # 药品大类
'foods' : set(), # 食物
'checks' : set(), # 检查
'departments': set(), # 科室
'symptoms' : set(), # 症状
}定义每个实体是从原数据的哪些字段中抽取出来
nodes_key_dict = {
'drugs' : ['common_drug', 'recommand_drug'], # 药品
'producers' : ['drug_detail'], # 药品大类
'foods' : ['not_eat', 'do_eat', 'recommand_eat'], # 食物
'checks' : ['check'], # 检查
'departments': ['cure_department'], # 科室
'symptoms' : ['symptom'] # 症状
}2.3. 定义关系
定义所有实体间的关系集
edges_set_dict = {
'acompany' : set(), # 疾病并发关系
'not_eat' : set(), # 疾病-忌吃食物关系
'do_eat' : set(), # 疾病-宜吃食物关系
'recommand_eat' : set(), # 疾病-推荐吃食物关系
'common_drug' : set(), # 疾病-通用药品关系
'recommand_drug': set(), # 疾病-热门药品关系
'production' : set(), # 厂商-药物关系
'check' : set(), # 疾病-检查关系
'belongs_to' : set(), # 科室-科室关系
'category' : set(), # 疾病与科室之间的关系
'symptom' : set(), # 疾病症状关系
}定义每个关系是从原数据的哪些字段中抽取出来的
edges_key_dict = {
'acompany' : ['name', 'acompany'], # 疾病并发关系
'not_eat' : ['name', 'not_eat'], # 疾病-忌吃食物关系
'do_eat' : ['name', 'do_eat'], # 疾病-宜吃食物关系
'recommand_eat' : ['name', 'recommand_eat'], # 疾病-推荐吃食物关系
'common_drug' : ['name', 'common_drug'], # 疾病-通用药品关系
'recommand_drug': ['name', 'recommand_drug'], # 疾病-热门药品关系
'production' : ['drug_detail', 'drug_detail'], # 厂商-药物关系
'check' : ['name', 'check'], # 疾病-检查关系
'belongs_to' : ['cure_department', 'cure_department'], # 科室-科室关系
'category' : ['name', 'cure_department'], # 疾病与科室之间的关系
'symptom' : ['name', 'symptom'], # 疾病症状关系
}定义每个关系的三元组
edges_tuple_dict = {
'acompany' : ['diseases', 'diseases', '并发症'], # 疾病并发关系
'not_eat' : ['diseases', 'foods', '忌吃'], # 疾病-忌吃食物关系
'do_eat' : ['diseases', 'foods', '宜吃'], # 疾病-宜吃食物关系
'recommand_eat' : ['diseases', 'foods', '推荐食谱'], # 疾病-推荐吃食物关系
'common_drug' : ['diseases', 'drugs', '常用药品'], # 疾病-通用药品关系
'recommand_drug': ['diseases', 'drugs', '好评药品'], # 疾病-热门药品关系
'production' : ['producers', 'drugs', '生产药品'], # 厂商-药物关系
'check' : ['diseases', 'checks', '诊断检查'], # 疾病-检查关系
'belongs_to' : ['departments', 'departments', '属于'], # 科室-科室关系
'category' : ['diseases', 'departments', '所属科室'], # 疾病与科室之间的关系
'symptom' : ['diseases', 'symptoms', '症状'], # 疾病症状关系
}2.4. 抽取数据
抽取 diseases 实体集
for data in tqdm(open(data_path, 'r', encoding='utf8')):
data_json = json.loads(data)
disease_info = {}
for key in diseases_nodes_attr:
disease_info[key] = data_json.get(key, '')
diseases_nodes.append(disease_info)抽取结果样例
抽取其他实体集
for data in tqdm(open(data_path, 'r', encoding='utf8')):
data_json = json.loads(data)
for node in nodes_set_dict:
for key in nodes_key_dict[node]:
if key in data_json:
# 特殊处理字段
if key == 'drug_detail':
nodes_set_dict[node] |= set([i.split('(')[0] for i in data_json[key]])
else:
nodes_set_dict[node] |= set(data_json[key])抽取结果样例
抽取关系集
for data in tqdm(open(data_path, 'r', encoding='utf8')):
data_json = json.loads(data)
for key in edges_set_dict:
p_key, q_key = edges_key_dict[key]
if p_key in data_json and q_key in data_json:
p, q = data_json[p_key], data_json[q_key]
if key == 'category':
if len(q) == 1:
edges_set_dict[key].add((p, q[0]))
elif len(q) == 2:
edges_set_dict[key].add((p, q[1]))
elif key == 'belongs_to':
if len(q) == 2:
big, small = q
edges_set_dict[key].add((small, big))
elif key == 'production':
edges_set_dict[key] |= set([(i.split('(')[0], i.split('(')[-1].replace(')', '')) for i in q])
else:
for _ in q:
edges_set_dict[key].add((p, _))抽取结果样例
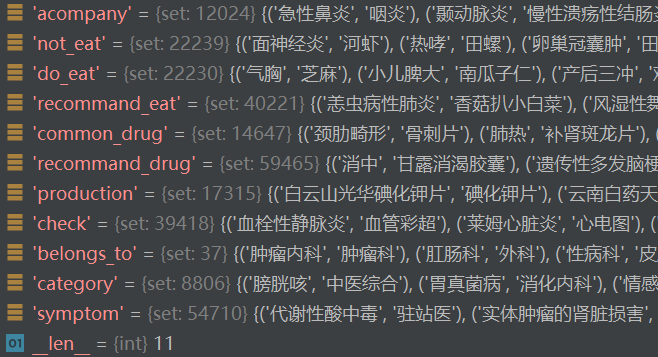
最后将实体保存,方便后序使用
tmp = {k: list(v) for k, v in nodes_set_dict.items()}
tmp['diseases'] = [_['name'] for _ in diseases_nodes]
with open('data/dict.json', 'w', encoding='utf8') as f:
json.dump(tmp, f, indent=4, ensure_ascii=False)3. 数据导入
连接neo4j数据库
# 老版本连接语句
graph = Graph(host="127.0.0.1", http_port=7474, user="neo4j", password="123456")
# 新版本连接语句
graph = Graph('http://localhost:7474', auth=('neo4j', '123456'))diseases 实体集入库
for disease_info in tqdm(diseases_nodes):
node = Node('diseases', **disease_info)
graph.create(node)其他实体集入库
for key, values in nodes_set_dict.items():
print(key)
for name in tqdm(values):
node = Node(key, name=name)
graph.create(node)关系集入库
for rel_type, edges in edges_set_dict.items():
start_node, end_node, rel_name = edges_tuple_dict[rel_type]
print(start_node, end_node, rel_name)
for edge in tqdm(edges):
p, q = edge
query = f"""
match(p:{start_node}),(q:{end_node})
where p.name='{p}' and q.name='{q}'
merge (p)-[rel:{rel_name}{{name:'{rel_name}'}}]->(q)
"""
try:
graph.run(query)
except Exception as e:
print(e)至此,知识图谱基础部分已经构建完毕